Qualitative und quantitative Analyse von Schweinefleisch in Rindfleisch mit der LC-MS Methode und der MRM-Technologie
Fleisch-Authentizitätsprüfung mit der Triple-Quadrupol-Massenspektrometer LCMS-8050 und Nexera X2 HPLC von Shimadzu
Lebensmittelbetrug ist ein Problem von globalem Ausmaß. Olivenöle werden verschnitten, Weine und Champagner gestreckt, Bio-Lebensmittel falsch bezeichnet, Produkte umetikettiert. Vor einigen Jahren wurde in Fertigprodukte statt des deklarierten Rindfleischs Pferdefleisch hinzugegeben. Solche Täuschungen wirken sich meist auf die Qualität und den Geschmack des Endprodukts aus; sie täuschen aber auch die Verbraucher und verursachen wirtschaftlichen Schaden. Wer an Allergien oder Unverträglichkeiten leidet, kann durch falsche Angaben über die Fleischherkunft sogar gesundheitliche Probleme bekommen. Den Verbrauchern wie auch den Industrien in den Lieferketten ist dieser Missbrauch zunehmend bewusst.
Allerdings ist der Nachweis bestimmter Fleischsorten bezogen auf ihre Herkunft bzw. Spezies problematisch. Gegenwärtig werden zur Authentifizierung der Spezies routinemäßig das antikörperbasierte Nachweisverfahren ELISA eingesetzt und PCR-Methoden (polymerase chain reaction). Diese Techniken bringen jedoch einige Nachteile mit sich. So führt die ELISA-Methode oft zu falsch positiven oder falsch negativen Ergebnissen. Eine PCR-Methode (ohne Verwendung der Real-Time PCR) ist nicht ohne Weiteres für die Quantifizierung geeignet und benötigt eine längere Analysezeit.
Aufgrund der Schwächen dieser Methoden ist die LC-MS Methode unter Verwendung der MRM-Technologie und der Aufnahme von charakteristischen Peptiden jeweiliger Spezies das Verfahren der Wahl für die qualitative und quantitative Analyse von Fleisch-Authentifizierungen.
Probenvorbereitung und Analyseaufbau
Zu Beginn wurde das Fleisch als Probenmaterial in Scheiben geschnitten, mit einem elektrischen Fleischwolf zerkleinert und anschließend unterschiedliche Mischungsverhältnisse für die Analyse hergestellt (Rindfleisch gespickt mit Schweinefleisch im Verhältnis von 5 %, 10 %, 20 %, 40 %, 60 % und 80 %). Etwa 2 g der jeweilig vorbereiteten Fleischproben wurden in einem Zentrifugenröhrchen (50 ml) eingewogen. Als Extraktionspuffer diente 7 M Harnstoff, 2 M Thioharnstoff und 50 mM Tris-HCl mit einem pH-Wert von 8 – der Probe hinzugefügt in einem Volumen von 10 ml. Nach 20 Sekunden Schütteln fand eine Homogenisierung aller Proben mittels einem Ultra Turrax T-25 statt.
Anschließend folgten Dispersionsschritte, 30 s bei 8.000 U/min, daraufhin 30 s bei 9.000 U/min und letztlich 30 s bei 11.000 U/ min. Für die anschließende Extraktion wurden die Proben 30 Minuten lang bei 4 °C und 20.000 U/ min zentrifugiert. Nachdem die Proben mit DTT und IAA zur Reaktion gebracht wurden, kam für den Proteinverdau Trypsin zum Einsatz. Die Probenvorbereitung abschließend, erfolgte eine Vorbehandlung der verdauten Proben mit einer HLB-SPE-Säule, um die Analyten für die Messung aufzukonzentrieren.
Für die Analytik zur Lebensmittelechtheit gehören massenspektrometrische Analysen in Kombination mit einer Flüssigkeits-Chromatographie zum Goldstandard. Insbesondere haben sich hier Triple-Quadrupol-Massenspektrometer bewährt. In dem hier vorgestellten Analysebeispiel wurde das Triple-Quadrupol-Massenspektrometer LCMS-8050 in Kombination mit der Nexera X2 HPLC von Shimadzu verwendet, ein applikationsspezifisches System verschiedenster technischer Ausprägung.
Kollisionsenergien-Optimierung für charakteristische Peptide
Die vier Aminosäurensequenzen bzw. Peptide SALAHAVQSSR, TLAFLFAER, YDIINLR und LVVITAGAR wurden vorab als spezifische Peptide für Schweinefleisch ermittelt. Dagegen zählen die Peptide TLALLFSGPASGEAEGGPK, EASGPINFTVFLNMFGEK, HPSDFGADAQAAMSK, ALEDQLSELK und LVIITAGARF als charakteristisch für Rindfleisch. Für die weitere Analyse optimiert die Software Skyline diese spezifischen Peptide nach der Kollisionsenergie (CE), um eine bestmögliche quantitative Methode zu etablieren (Abb. 2 und 3).
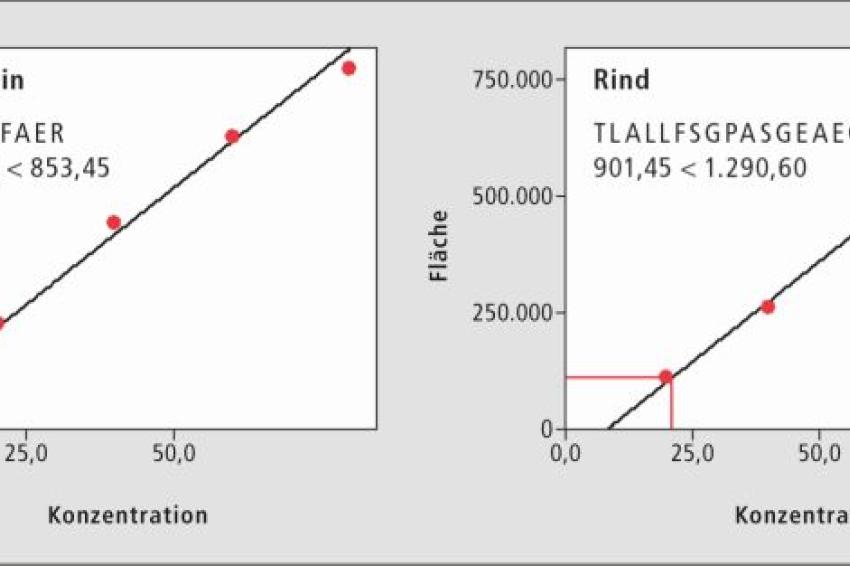
Eine ideale Wahl der Kollisionsenergien in dem zweiten Quadrupol (Kollisionszelle) ist entscheidend für die induzierte Stoß-Fragmentierung der im ersten Quadrupol selektierten Zielpeptide. Eine richtige Einstellung der Kollisionsenergie wirkt sich auf die Art bzw. Struktur aus sowie auf die Sensitivität der Produkt-Ionen, die letztlich im dritten Quadrupol ausgelesen werden. Unter Berücksichtigung dieser so ermittelten Optimierungsergebnisse und der hier gegebenen Matrix-Effekte durch das Probenmaterial, wurden die charakteristischen Peptide TLAFLFAER (m/z 534,30 > 853,45) und TLALLFSGPASGEAEGGPK (m/z 901,45 > 1290,60) für die Quantifizierung der Fleischproben von Schwein und Rind ausgewählt.
Erstellung der Kalibrierkurve für Schweine- und Rindfleisch
Um den Gehalt der verschiedenen Anteile der beiden Fleischsorten in den Proben Schwein (im Anteil 5 %, 10 %, 20 %, 40 %, 60 % und 80 %) und Rind (im Anteil 20 %, 40 %, 60 %, 80 %, 90 % und 95 %) zu bestimmen, wurden zwei Regressionsgeraden der jeweiligen Spezies erstellt. Das Ergebnis zeigte, dass die Genauigkeit für die Kalibrierkurve für Schweinefleisch bei 87,4~110,2 % und für Rindfleisch bei 95,2~104,9 % liegt. Der Korrelationskoeffizient (r-Wert) der Geraden beträgt 0,9960 und 0,9982 für das entsprechend ausgewählte Marker-Peptid des Schweine- und Rindfleischs.
Fazit
Die qualitative Bestimmung der Fleischsorten konnte durch die Auswahl von vier charakteristischen Peptiden der jeweiligen Spezies realisiert werden. So wurden die Aminosäuresequenzen SALAHAVQSSR, TLAFLFAER, YDIINLR und LVVITAGAR spezifisch für Schweineifleisch ermittelt sowie TLALLFSGPASGEAEGGPK, EASGPINFTVFLNMFGEK, HPSDFGADAQAAMSK, ALEDQLSELK und LVIITAGAR kennzeichnend für Rindfleisch.
Des Weiteren ermöglicht die massenspektrometrische Analyse eine Quantifizierung verschiedener Fleischanteile unterschiedlicher Spezies in einem Probengemisch. Durch die Kompatibilität der Shimadzu Software Labsolutions mit der Open-Source-Software Skyline ließen sich die detektierten Peptide direkt im weiteren Verlauf der MS-Analytik optimieren. So konnten bequem die Kollisionsenergien als wichtiger Parameter zur Quantifizierung angepasst werden.
Als Marker für Schweinfleisch dient hier das Peptid TLAFLFAER bei m/z 534,3 mit dem dazugehörigen MRM-Übergang von m/z 853,45 und für Rindfleisch das Peptid TLALLFSGGPASGEAEGGPK bei m/z 901,45 mit dem entsprechenden MRM-Übergang von m/z 1290,6. Das Resultat zeigt eine Kalibrierkurve mit einer Genauigkeit von 87,4~110,2 % und einen r-Wert von 0,9960 für den Schweinefleischanteil und eine Genauigkeit von 95,2~104,9 % und einem r-Wert von 0,9982 für den Anteil des Rindfleisches in der Probe.
Die Methode zeigt die Möglichkeit für die Analytik in der Nahrungsmittelkontrolle und zur Identifizierung bzw. Authentifizierung verschiedener Fleischsorten anhand der charakteristischen Aminosäuresequenzen. Durch die Weiterentwicklung dieser Methode ist zu erwarten, dass auch zusätzliche Fleischsorten anderer Spezies wie bspw. Geflügel mit aufgenommen werden können. So könnte innerhalb einer massenspektrometrischen Analyse das Spektrum der verwendeten Fleischsorten überprüft werden.